本周主要做了以下工作:
阅读两篇 UC Berkeley 计算成像实验室的论文:
Antipa N , Kuo G , Heckel R , et al. DiffuserCam:lensless single-exposure 3D imaging [J]. Optica, 2018.
学习 上面两篇论文的 Diffuser Cam源码 DiffuserCam
Paper 1 Title 《漫射相机:无镜片单曝光三维成像》
Antipa N , Kuo G , Heckel R , et al. DiffuserCam:lensless single-exposure 3D imaging [J]. Optica, 2018.
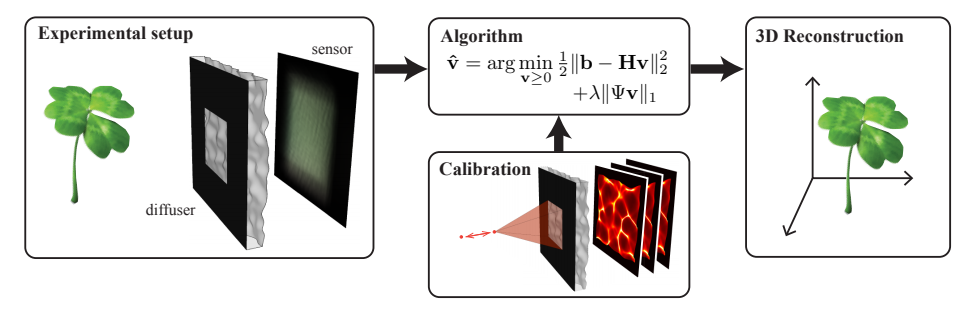
Contents 作者提出了一种基于diffuser (漫射片,毛玻璃片)的无物镜三维成像系统,由于每一个空间位置的点光源都能够通过diffuser产生伪随机折射 最终在sensor上呈现出一种 caustics(焦散线) 的图案,在数学上 称为Point Spead Functions (PSFs) ,便能够通过一些标定手段获得 diffuser对于三维空间中每一个空间点对应到sensor上一个图案的传递函数(矩阵),结合压缩感知 和最优化算法,进而实现通过混杂的二维sensor图案反演得到三维原图像。
本文针对大规模的(100million)像素块标定过程,引入了shift invariance assumption(位移不变性假设)和paraxial approximation(近轴近似),将标定规模降低至仅需标定 0轴 的范围,大大降低标定复杂度。
实验器材仅需:diffuser、sensor、aperture(用来控制diffuser成像投影在sensor上的大小)
大致的实验模型如下:在diffuser前加一个aperture,后方放置sensor 即可
通过 z轴 标定后得到H矩阵,应用最优化算法即可得到三维成像结果。
Specifics 设 sensor上图案矩阵的向量表示为 $\mathbf b$,diffuser的传递矩阵为 $\mathbf H$,三维场景元素矩阵的向量表示为 $\mathbf v$,则 有:
式中,$\mathbf H$ 的每一列都代表 三维场景中单个像素块对sensor的所有元素的贡献。
需要求解的问题,即:
为了应用压缩感知,需要 $\mathbf H$ 满足
每列都尽量不相关
焦散图案需要较高的空间频率
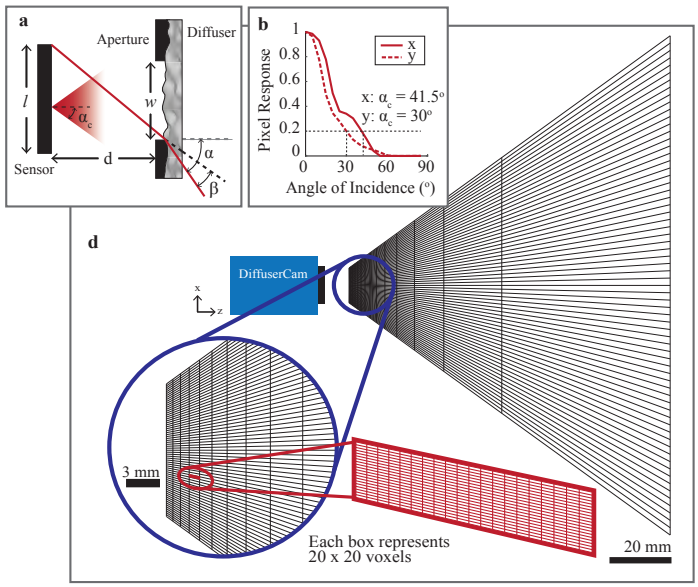
本实验装置有两个特性参数需要控制:
焦比 f-number(焦距与光圈比值):决定了成像分辨率
平均焦距:决定了最佳放置sensor的位置
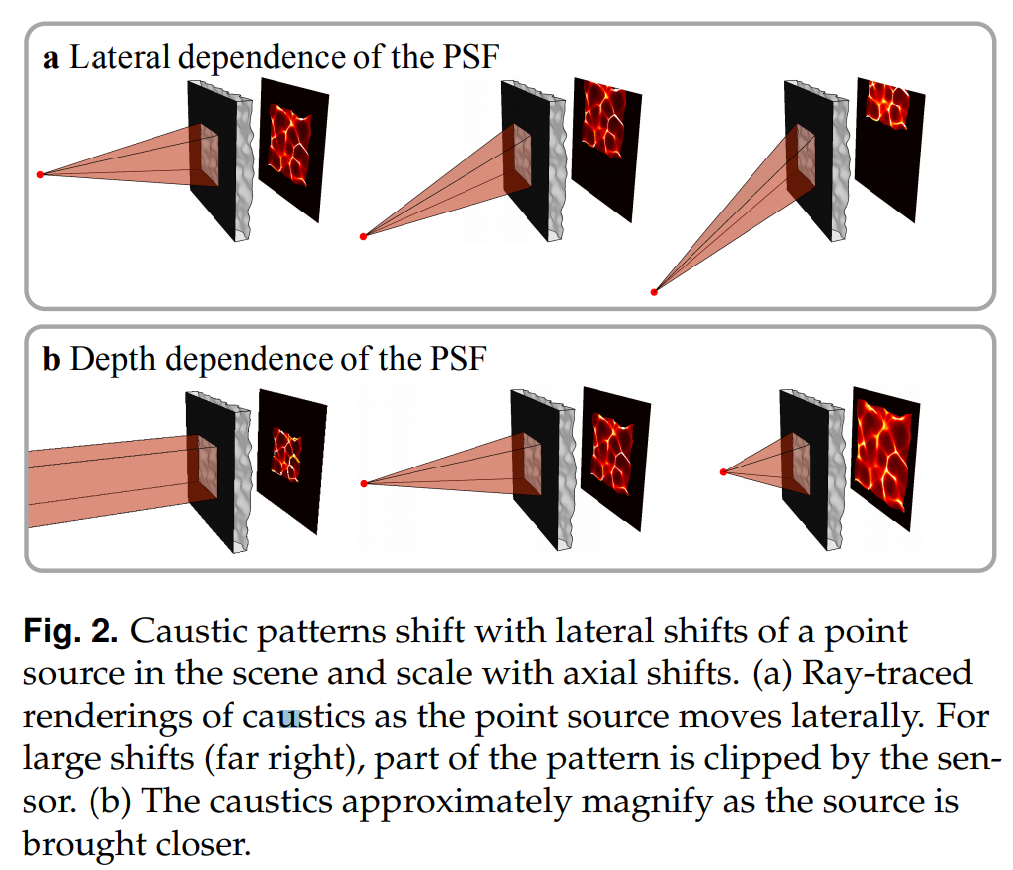
此外,还需要考虑的问题是 轴向位置的影响、侧向位移的影响
此处,在计算矩阵乘法 $\mathbf {Hv}$ 时,写成求和式,如下:
计算复杂度过高,从而引入了shift invariance assumption(位移不变性假设)和paraxial approximation(近轴近似),将同一 z轴坐标的 不同 $(x,y)$ 像素值产生的对sensor的成像近似为 正比于 同一z轴坐标 在$(0,0)$处产生的对sensor的成像,这样一来,上式的计算可以消掉两个维度:
$h(x’,y’;x,y,z) = h(x’+mx,y’+my;z)$
从而有:
其中,$*$ 表示求三维离散卷积,这种模型为本文提出的 convolution model,它拥有一些优点:
使得计算每 $\mathbf {Hv}$ 变得更简单
它提供了一个本系统满足稀疏性的一个有效的证据
使得标定过程变得更简单
在求解这样一个最优化模型时,由于规模太大,梯度投影计算复杂度过高,因此作者应用了针对大规模的ADMM算法(交替方向乘子法) (详情见2011年论文 S. Boyd, N. Parikh, E. Chu, B. Peleato, and J. Eckstein, “Distributed optimization and statistical learning via the alternating direction method of multipliers ,” Foundations and Trends in Machine Learning 3, 1–122 (2011).),因此需要变量分离 操作(查了一下,ADMM是双变量迭代优化算法):
经过分析,总的计算复杂度为 $O(n^3logn)$,受制于三维FFT复杂度。
此外,本文还提到了成像角度问题,最终设置了x和y方向的两个满足 两点分辨率 的临界角度 $41.5°,30°$
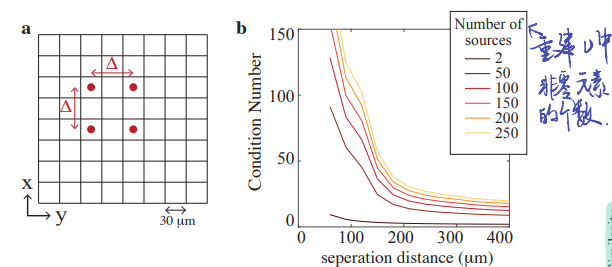
同时,作者提出了一种新的分辨率判别形式用来:multi-point resolution,即多点分辨率,此处是16个点。因为作者发现 本实验装置的重建分辨率与成像物体的复杂性有关 ,复杂的物体重建,多点分辨率设定会比两点的更优。
针对分辨率问题,作者提出了 Local condition nuber theory(局部条件数理论) ,即 定义局部条件数 为成像的 $\mathbf v$ 中非零元素的数量,显然,非零元素越多,效果越差(混杂现象多)。
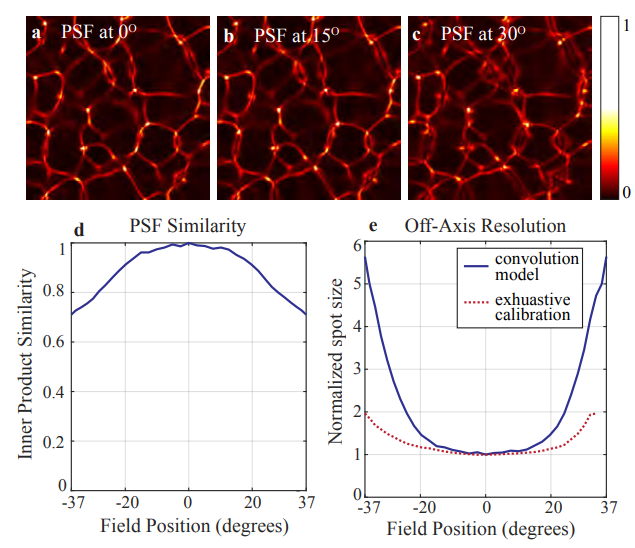
在实验阶段,作者为了验证近轴近似的正确性,在同一z轴坐标下,测出了不同角度的PSF图像 ,并将所有角度的PSF与0°的PSF作内积 ,并对比,得到图像:
理论上来讲,同意z轴坐标下所有角度的PSF都是相同的,从上图的结果来看,所有角度的PSF最差也有75%的相似度,因此可以认为近似成立。
上图右下角的子图中,蓝色实线为所有重建的单点峰宽与重建0°点的峰宽比值 ,并用0°点峰宽做标准化,红色点线为实行完全标定的结果(误差很小),然而为了降低复杂度,本实验仍然使用简化的标定方法。
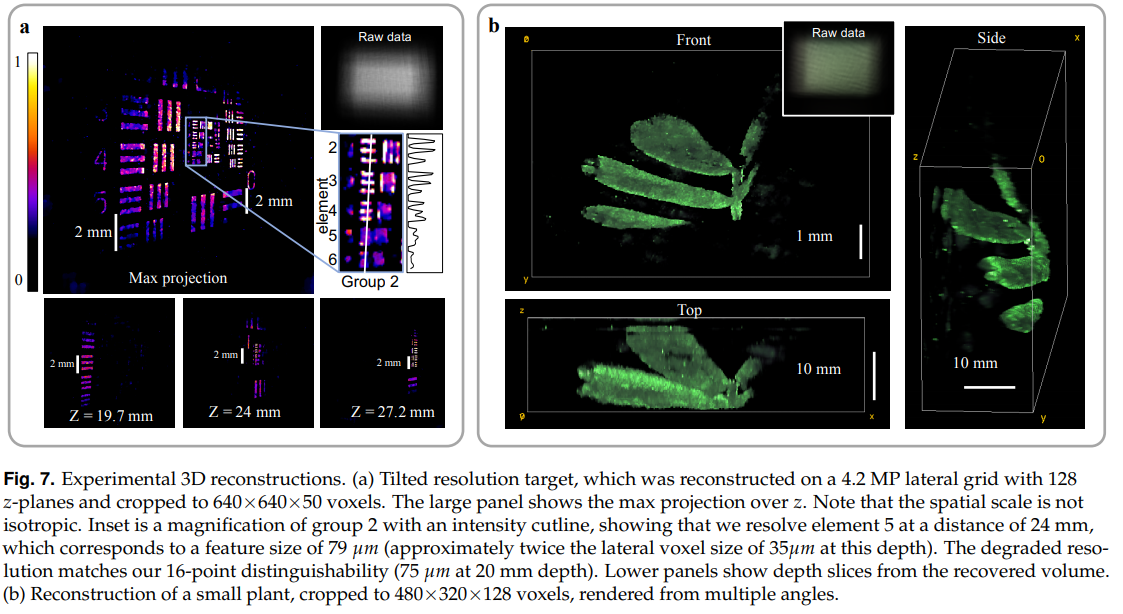
Experiments & Results 实验采用128个z轴平面,z=10.86mm ~ 36.26mm,三维图像在 $2048\times 2048\times 128$ 网格上重建,重建物体为USAF1951分辨率板 和一株绿色植物 ,结果如下:
图像处理知识点 关于cv2和matplotlib的imshow区别
建议尽量不要混用 ,如果非要的话,可以用以下方式处理:
'lena.jpg' )
关于fft计算线性卷积 和 (i)fftshift的作用 在数学与信号处理种,通常计算信号的卷积 是 将其通过傅里叶变换转换到频域,然后计算频域的乘积,再逆变换回去,DFT的快速算法是FFT,时间复杂度$O(nlogn)$,其具体实现见 我的blog:FFT及其优化 ,然而,可以证明FFT计算得到的其实是循环卷积 ,并不是我们通常想得到的线性卷积 。
为了解决上述问题,需要再傅里叶变换前进行一些操作,称之为padding 0(在卷积神经网络中又学到过,即 补零操作),之后再进行FFT就能得到线性卷积(可以严格证明)。
为了将频谱零频点移动到图像中央,我们需要对原图进行 (i)fftshift 操作
上述的 一半尺寸 指:偶数$\frac n 2$,奇数$\frac{n-1}2$
因此,在求卷积之前,我们都需要进行 (i)fftshift 操作
FISTA Algorithm FISTA(快速迭代阈值收缩算法)基于梯度下降,收敛复杂度为$O(1/k^2)$,比传统的ISTA(复杂度 $O(1/k)$)更快。
参考UC Berkeley计算成像实验室代码:
code
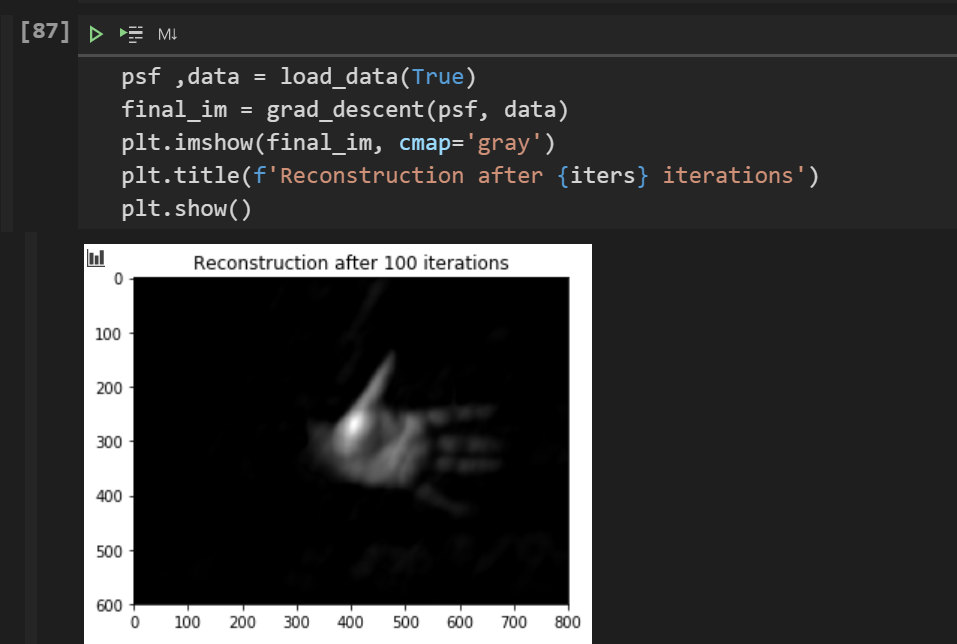
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 118 119 120 121 122 123 124 125 126 127 128 129 130 131 132 133 134 135 136 137 138 139 140 141 142 143 144 145 146 147 148 149 150 151 152 153 154 155 156 157 158 159 160 161 162 163 164 165 166 167 168 169 170 171 172 173 174 175 176 177 178 179 180 181 182 183 184 185 186 187 188 189 190 191 192 193 194 195 196 197 198 199 200 201 202 203 204 205 206 207 208 209 210 211 212 213 214 215 216 217 218 219 220 221 from PIL import Imageimport matplotlib.pyplot as pltimport numpy as npimport numpy.fft as fftfrom IPython import displayimport cv21 /8 'psf_sample.tif' 'rawdata_hand_sample.tif' 100 def load_data (show_im=True ):open (psf_data)open (raw_data)'float32' )'float32' )print (f"origin_size_psf: {psf.shape} " )print (f"origin_size_rawdata: {data.shape} " )5 :15 ,5 :15 ])def resize (img,factor ):int (-np.log2(factor))for i in range (num): 0.25 * (img[::2 ,::2 ,...]+img[1 ::2 ,::2 ,...]+img[::2 ,1 ::2 ,...]+img[1 ::2 ,1 ::2 ,...])return imgprint (f"new_size_psf: {psf.shape} " )print (f"new_size_data: {data.shape} " )if show_im:12 ,8 ))1 ,2 ,1 )'gray' )'psf' )1 ,2 ,2 )'gray' )'raw_data' )return psf,datadef nxt2 (x ):return int (2 **np.ceil(np.log2(x)))def init_mat (h ):max (h) + np.min (h)) / 2 2 *n-1 ) for n in h.shape]0 ] - h.shape[0 ]) // 2 0 ]1 ] - h.shape[1 ]) // 2 1 ]'ortho' )def crop (X ):return X[starti:endi,startj:endj]def pad (v ):return vpadreturn H,HH,v,utilsdef grad (AH, H, vk, b, crop, pad ):return np.real(calcAHerm(AH, diff, pad))def calcA (A, vk, crop ):return crop(fft.fftshift(fft.ifft2(A * Vk)))def calcAHerm (AH, diff, pad ):return fft.fftshift(fft.ifft2(AH * X))def grad_descent (h, b ):0 ],utils[1 ]1.8 /(np.max (HH * H)))0 def non_neg (xi ):0 )return xi1 for iterations in range (iters):if iterations % 10 == 0 :'gray' )f'Reconstruction after iteration {iterations} ' )True )return proj(crop(vk))def gd_update (vk, parent_var ):return vkdef nesterow_udpate (vk, p, mu, parent_var ):1 + mu) * preturn vk, pdef fista_update (vk, tk, xk, parent_var ):1 + np.sqrt(1 + 4 * tk * tk)) / 2 1 ) / tk1 *(xk - xk1)return vk, tk, xk'gray' )f'Reconstruction after {iters} iterations' )
成像结果: