周报:2020-11-30~2020-12-6
Paper 1
Title
《基于非负矩阵分解数值方法的集成光子腔光谱技术》
[1] Huang Z , Xiong D , Zhang G , et al. Nonnegative Matrix Factorization Numerical Method for Integrated Photonic Cavity Based Spectroscopy[J]. Journal of Nanomaterials, 2014, 2014:1-5.
Contents
数学模型与传统的光谱重建方法相同,最终都归结为一个解欠定线性方程组的问题,本文对于传递矩阵$T$的处理方式有所不同,它使用了线性代数方法 非负矩阵分解
这样一来,一个大规模的欠定问题(Undetermined)就被降维了,因为一个$n\times m$的矩阵被标示位了一个$n\times r$和一个$r\times m$的矩阵的乘积,类似于接下来要看的压缩感知
所选取的128个通道的滤波器如下图所示:
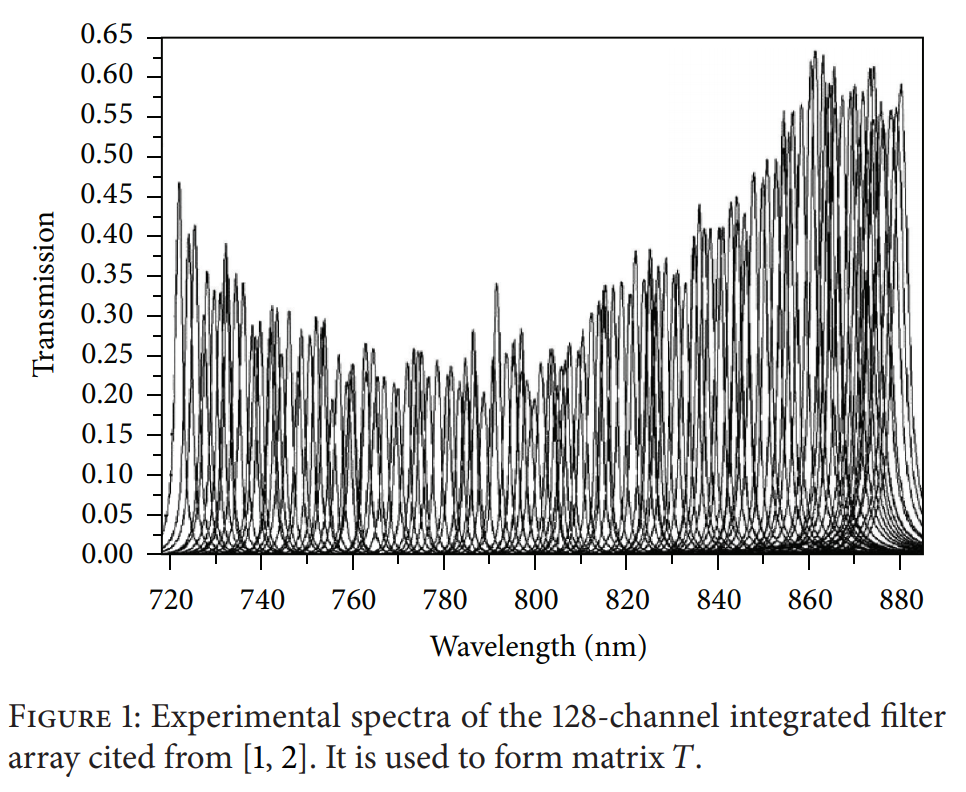
最终的解析解如下:

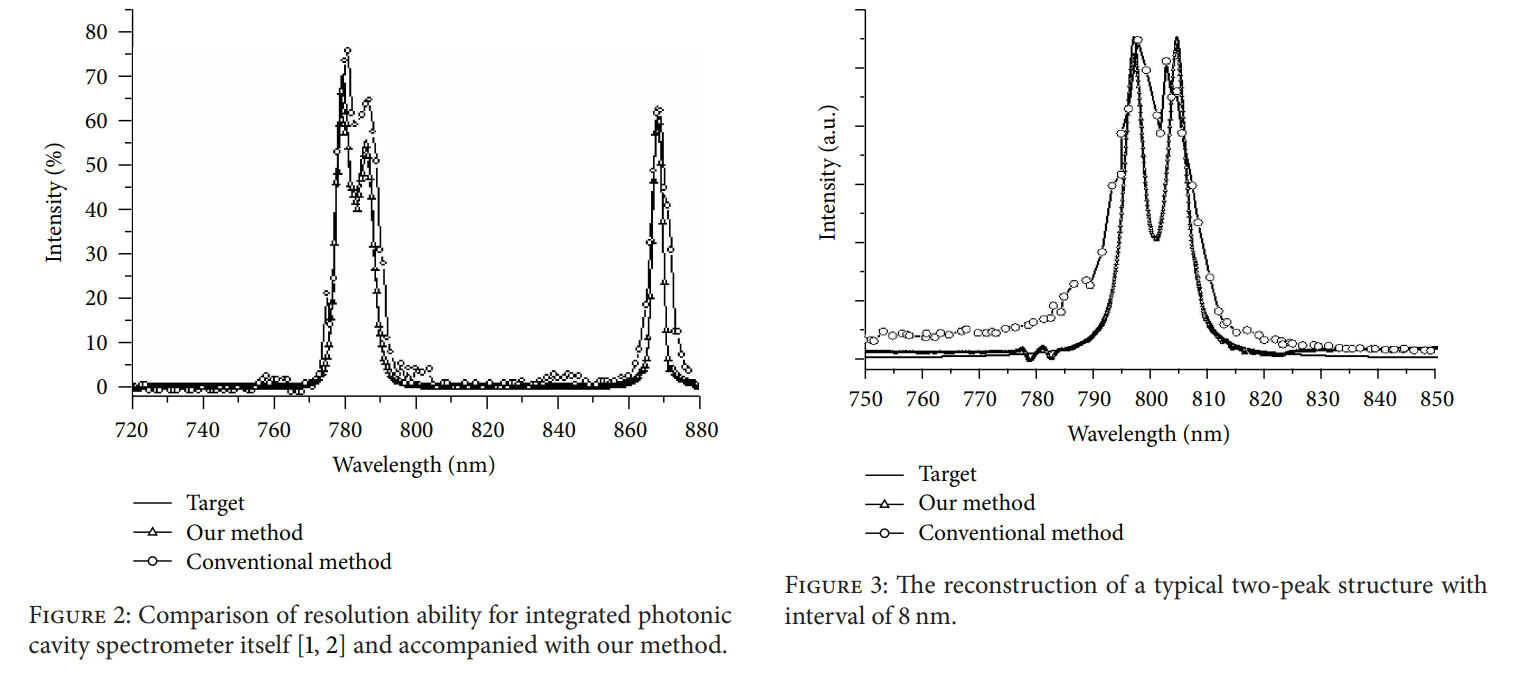
Results
重建结果要明显优于传统方法
Paper 2
这是一篇压缩感知经典文献,但是由于时间关系并没有看完,打算下周看完,并放在下周记录
OMP algorithm
这周还完成了一个比较经典的压缩感知重建算法OMP算法(正交匹配追踪), 它是传统的MP算法的改进
参考资料:
[1] Pati Y C , Rezaiifar R , Krishnaprasad P S . Orthogonal Matching Pursuit: Recursive Function Approximation with Applications to Wavelet Decomposition[C]// 1993:40-44.
正交匹配跟踪算法OMP - 阿珺的文章 - 知乎 https://zhuanlan.zhihu.com/p/52276805
形象易懂讲解算法II——压缩感知 - 咚懂咚懂咚的文章 - 知乎 https://zhuanlan.zhihu.com/p/22445302
Contents
对于一个压缩感知问题,我们可以写成如下的一个矩阵表示的线性方程组形式
目前,已知向量$y$和压缩感知矩阵$A$,需要求向量$x$,令$y$的长度位$n$,$x$的长度为$m$,则满足$n<<m$,是一个典型的欠定问题
我们认为$A$的每一列元素都是一个基信号,称为原子,可以将A表示为
于是,我们可以将$y$写成每一个$A$原子的线性组合:
OMP算法流程:
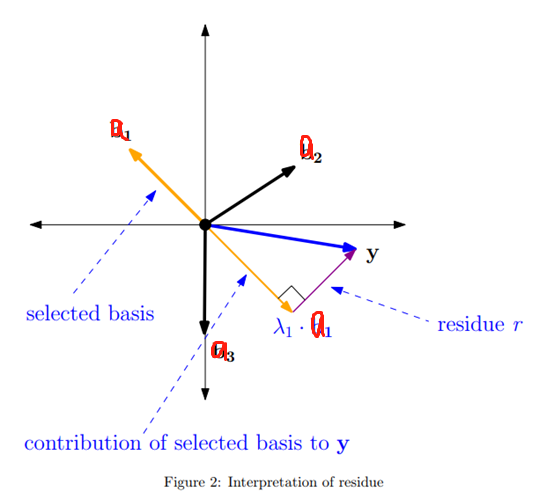
定义一个残差值 $r$(初值为$y$),一个空的原子序列$A_{new}$,和一个空的稀疏系数值$x_{rec}$
计算A中每个原子对于残差$r$的贡献,即 计算 $\langle a_i,r\rangle$,并找到对y影响最大的原子,即 绝对值最大的原子:
- 将这个最大的贡献值定义为A与y的相关系数,并将这个原子加入原子序列中
- 计算该原子序列对于y的贡献,得到的值用于更新$x_{rec}$
- 从y中减去这个原子序列贡献的影响,得到新的残差,即 从y中减去所有与该原子序列有关的信息,使得新残差r与该原子序列正交,如下图所示:
- 回到第2步,反复迭代,直到达到迭代次数 $I$(即 稀疏系数$K$)
最终得到的$x_{rec}$即为我们需要的稀疏系数向量
需要注意的是:OMP算法要保证压缩感知矩阵A的每个列(原子)都两两不相关,否则容易出现错误的解
Experiment
这次用到的是一个$256\times256$的lena.jpg,重建单通道(Blue)像素值
采用DCT(离散余弦变换)作为系数基矩阵,随机的高斯分布为观测矩阵
结果如下(其中,k为稀疏系数,sample_rate为采样率)


Code
code
1 | |
本博客所有文章除特别声明外,均采用 CC BY-SA 4.0 协议 ,转载请注明出处!